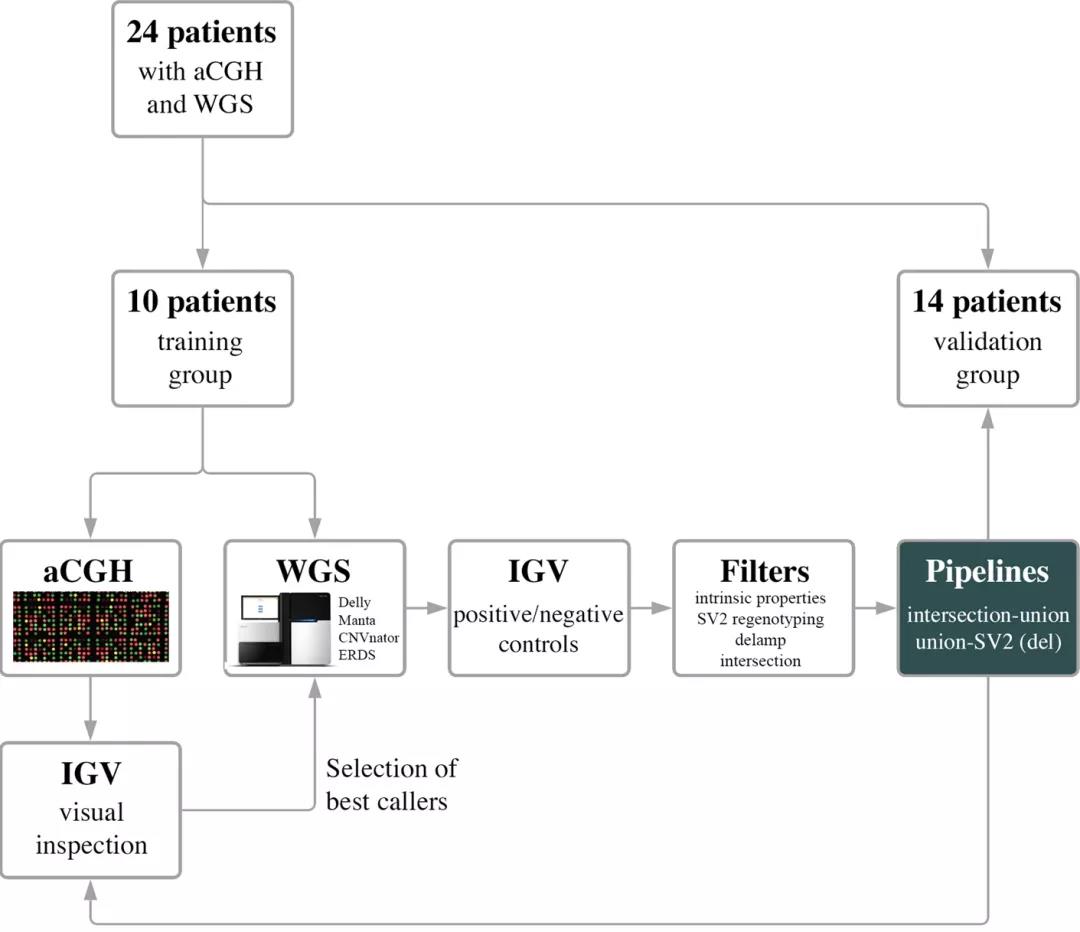
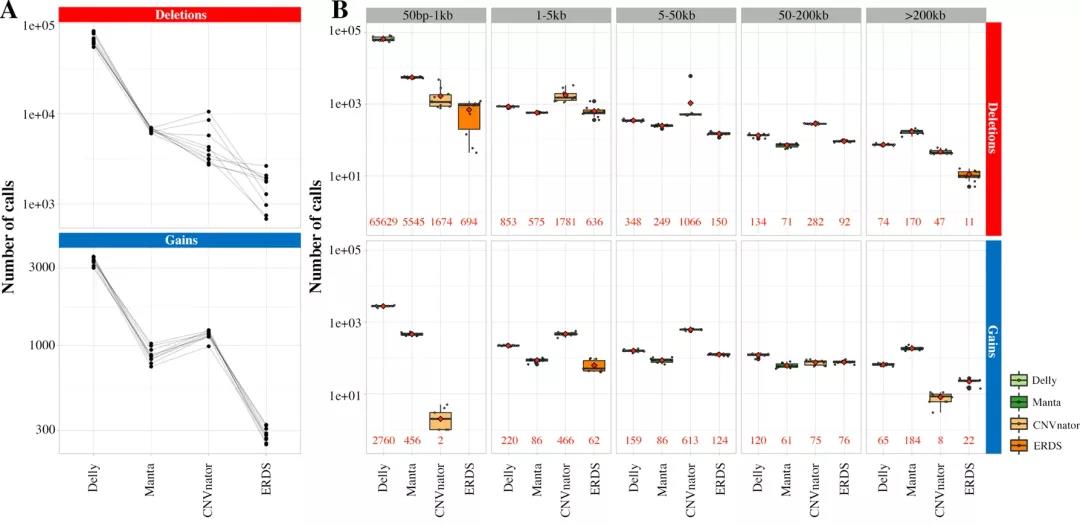
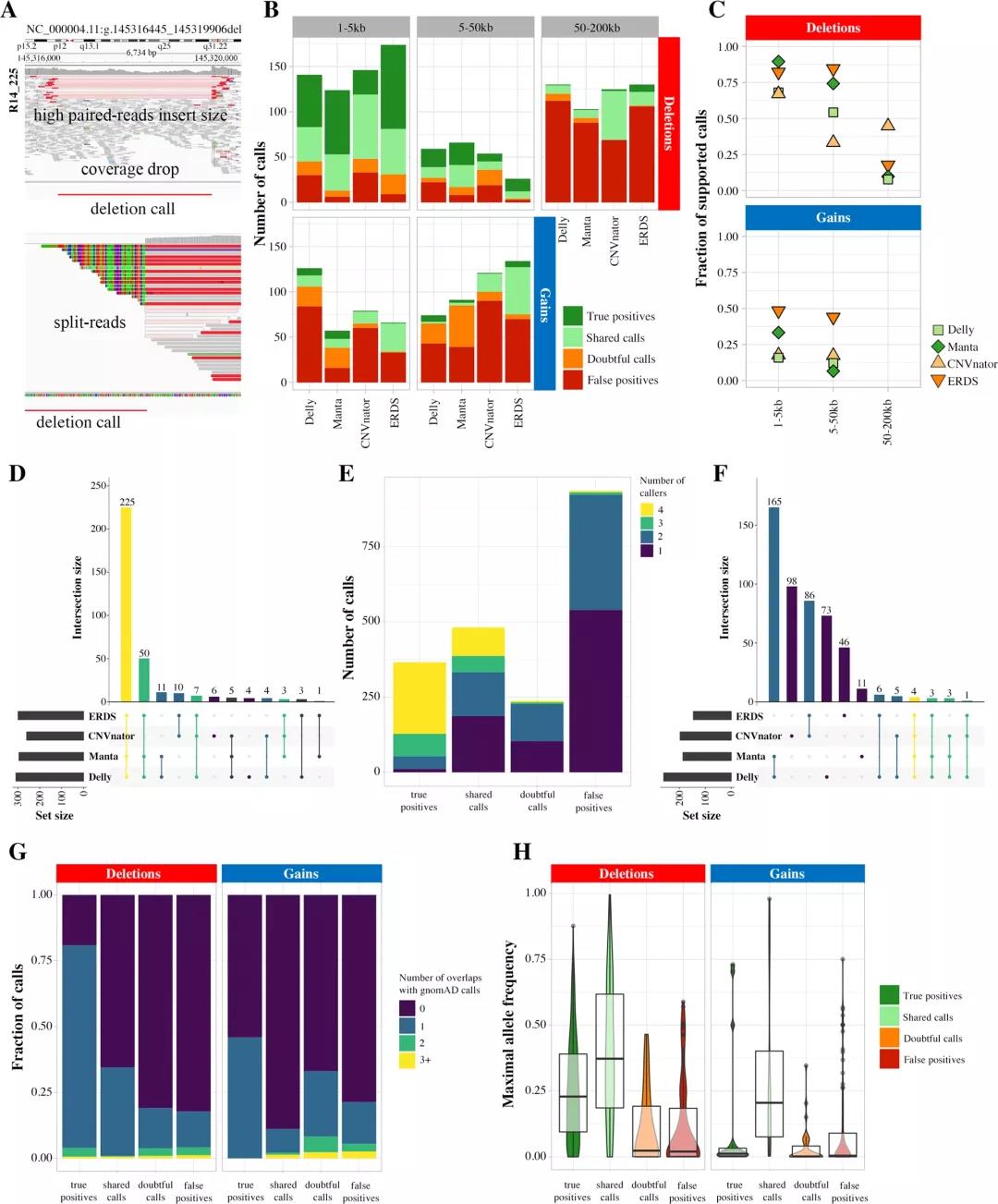
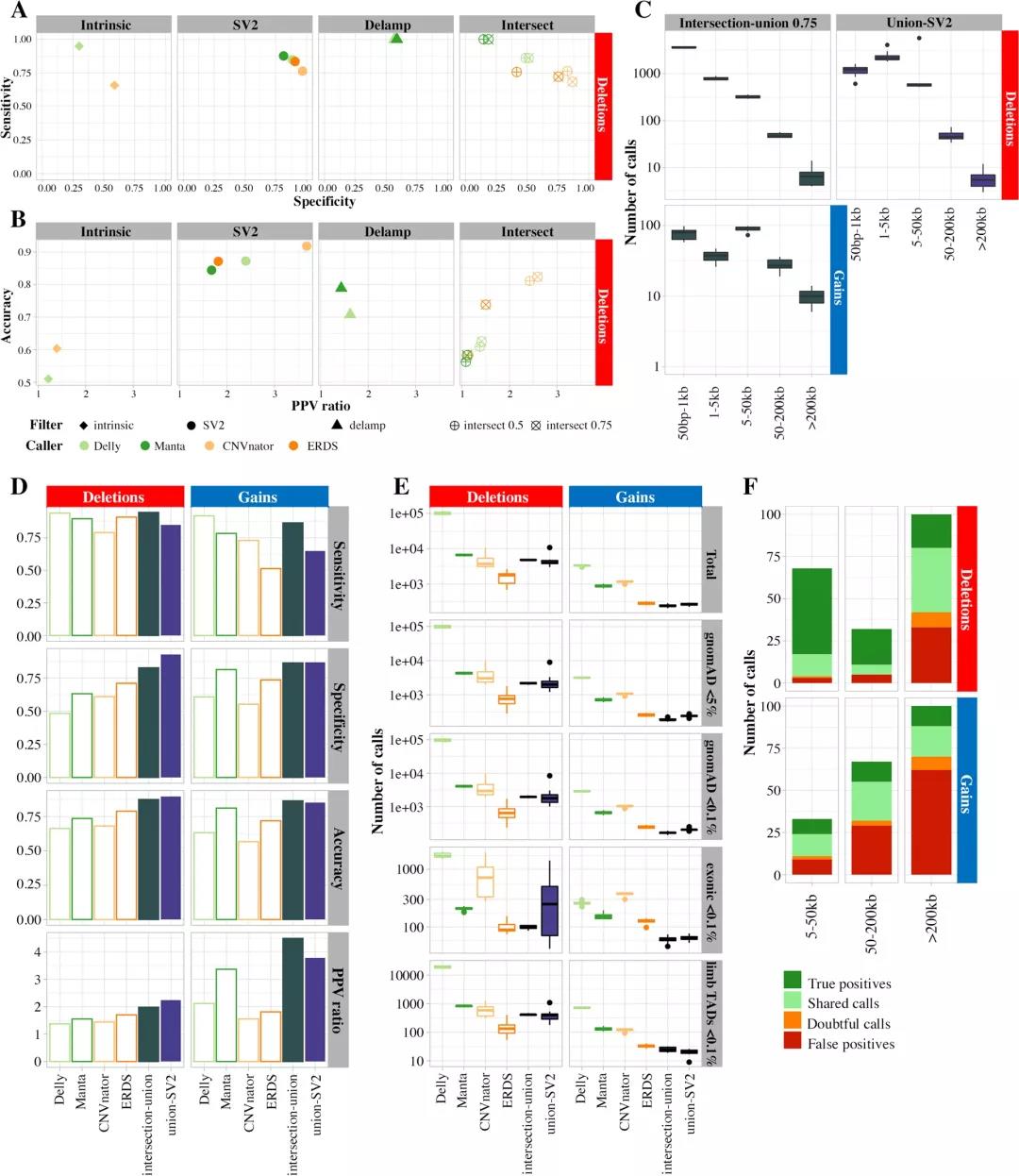
背景 拷贝数变异(CNVs)是大于50个碱基对的缺失、重复或插入,在人类基因组变异中占很大比例,对人的身体健康有很大影响。目前,基于芯片的检测方法在临床中广泛应用,但全基因组测序(WGS)有望同时检测CNVs和更小的变异,因此,利用WGS数据准确的检测CNVs在临床检测中至关重要。目前,基于WGS数据检测CNV的算法原理大致分为:paired-end reads, split reads和coverage depth,文章通过4种基于不同算法原理的CNV检测软件(Manta, Delly, ERDS, CNVnator)和一个基因分型工具(SV2)结合,得到可靠的CNVs结果。 分析方法 文章从24例肢体畸形患者中随机抽取10例作为训练组。对10例样本进行aCGH检测CNV并通过IGV确认,同时基于~30X WGS数据,选择4个效果较好的CNV鉴定工具(Manta, Delly, ERDS, CNVnator)进行下一步的分析。通过不同软件的组合与过滤获得最终检测结果,确定结果的准确性,并通过剩余的14例样本进行方案验证(图 1)。 图 1. 样本选择与分析流程 对于4种CNV检测软件,Delly与Manta的原理主要是基于paired-end检测,CNVnator与ERDS的原理主要是基于coverage depth检测,文章对CNV检测结果进行过滤与合并:对于相同原理软件的检测结果,如果CNV区域存在75%的交集,检测到不同类型的CNV则删除结果,检测到相同类型的CNV则合并结果;不同原理软件之间的结果,交集的区域调整为50%;再通过SV2进行基因分型,并对所有CNV结果进行比较过滤(图 2)。 图 2. CNV结果分析流程 分析结果 4种软件的检测结果差异较大。其中基于paired-end的软件检测到更多的CNV结果,尤其在50bp-1kb的缺失中差异明显;CNVnator对1-50k范围内的检测则更加敏感;相同检测原理的软件之间检测结果一致性相对较高;Delly和CNVnator相比于Manta和ERDS软件更加敏感,而Manta和ERDS的检测结果在大约一半的病例中得到相互验证(图 3)。 图 3. 4种软件的检测结果 对检测结果随机选择1278个缺失和748个插入进行IGV查看验证,软件检测结果与真实的CNV区域重叠范围为6.6%-89.5%之间,小的缺失型CNV比插入和大的缺失型CNV更易被检出。ERDS和Manta软件对1-50 kb缺失型CNV的检测更准确;Delly和CNVnator软件对1 ~ 5 kb缺失型CNV的真阳性率达到50%以上;ERDS对大片段CNV检测的敏感性最高;超过50kb的插入型CNV在基于coverage depth检测的软件中敏感性较高,但是此类CNV通过IGV查看时都没有得到验证。验证结果显示,假阳性的CNV大多只由一种软件检出,多种软件中共同检出的结果基本可以确保真实性,而且大多真实的结果能够通过genomAD数据库得到验证(图 4)。 图 4. 不同软件对不同类型CNV的检测结果及IGV验证 通过366个真实的CNV(329个缺失,37个插入)和940个假阳性CNV(505个缺失,435个插入)对CNV过滤方法的准确度进行评估。结果表明,根据4种软件的结果对CNVs进行过滤,对真实的灵敏度没有影响,而且可以增加准确性;SV2基因分型对缺失表现良好,但对插入的敏感性较低(图 5)。 图 5. 4种软件结合与SV2过滤的CNV结果。 结论 利用WGS数据检测CNV可以解决aCGH中检测区域局限、断点不明确的问题,但传统的检测方法准确性不高,而使用高深度或长读长测序的成本太高,不适用于临床检测。文章中使用多种CNV检测工具进行过滤的方法很好的解决了上述问题,4种检测软件依据不同的算法原理检测CNV,结合不同软件结果可以有效提高检测的准确性,为WGS数据检测CNV的临床应用开辟了新思路。 参考文献 Coutelier, M., Holtgrewe, M., Jäger, M. et al. Combining callers improves the detection of copy number variants from whole-genome sequencing. Eur J Hum Genet (2021). https://doi.org/10.1038/s41431-021-00983-x.