

背景
单倍剂量不足指一个等位基因突变或者缺失后后,另一个等位基因能正常表达,但这种基因表达翻译后的蛋白水平只有正常的50%,不足以维持正常的生理功能,导致特定表型出现。导致单倍剂量不足的原因有多种,比如一个基因的拷贝发生缺失,或者突变导致不能产生正常的mRNA,或者特殊情况下mRNA或蛋白质不稳定导致降解等。单倍剂量不足现象是导致遗传病发生的一个原因。哪些基因会发生单倍剂量不足呢?目前单倍剂量不足数据来源主要有3个方面:基于疾病的研究、生信软件预测和高通量筛查。
/
一、基于疾病的研究
最直接的数据来源便是基于疾病的研究,典型的数据库为OMIM数据库。研究报道中显示,在筛选出的299个人类单倍型剂量不足的基因中,有88个基因只在OMIM数据库中显示;94个基因只在文献中进行了报道;另外117个基因在OMIM与文献中显示了一致的结果。此外,有多个疾病数据库都对单倍剂量不足有所记载(表1)。
表1. 搜集单倍剂量不足的数据库

二、生信软件预测
基于疾病的研究获得的数据库记录范围有限,为了满足基因检测的需要,使用一种覆盖更全面的方法很有必要,因此从生物信息学的角度便产生了相应的预测软件。
最早单倍剂量不足预测工作的文献发表于2010年,基本流程为:从数据库中获取相关基因及其特性;根据数据库中搜集的存在单倍剂量不足的基因信息构建训练模型;使用该模型扫描基因组中蛋白编码基因预测单倍剂量不足基因(图1)。
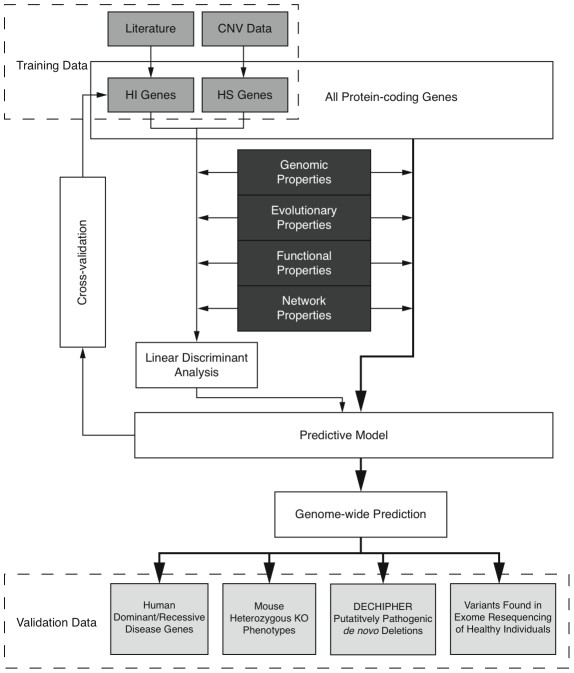
图1. 单倍剂量不足基因预测模型
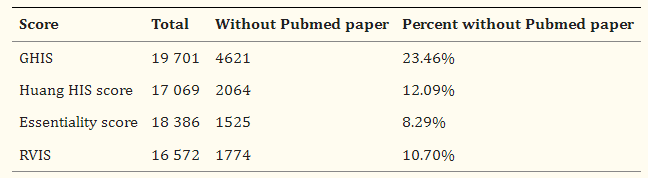
目前,已有多种单倍剂量不足预测方法,通过这些方法发现了很多潜在的相关基因(表2)。
表2. 单倍剂量不足基因预测模型
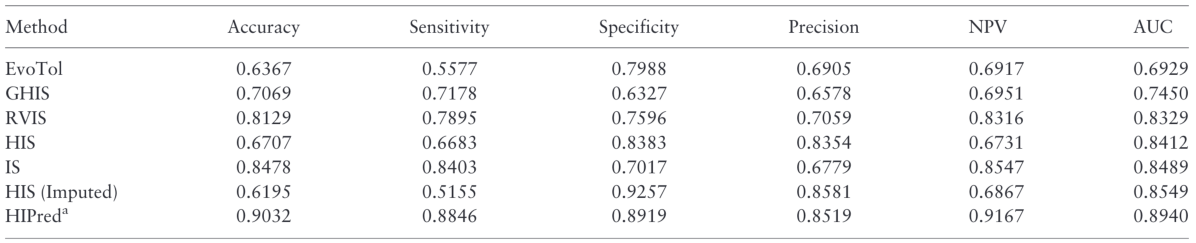
HIPred软件的作者比较了7款相关软件,结果显示,该软件各项指标都显示了最好的性能(表3)。
表3. 七款单倍剂量不足基因预测软件性能比较
三、高通量筛查
模型预测的方法可以发现一些单倍剂量不足的基因,但是也存在一定的不足:预测结果受训练模型数据库信息的影响。因此,高通量筛查的方法得到了广泛应用,常用技术手段为CRISPR。已有研究单位通过此方法对所有人类已知的基因进行了研究。基本流程为:通过CRISPR对单倍体细胞文库的指定基因进行敲除;敲除后的细胞文库与另一个未进行敲除的单倍型细胞文库融合;进行细胞培养并检查细胞活性(图2)。
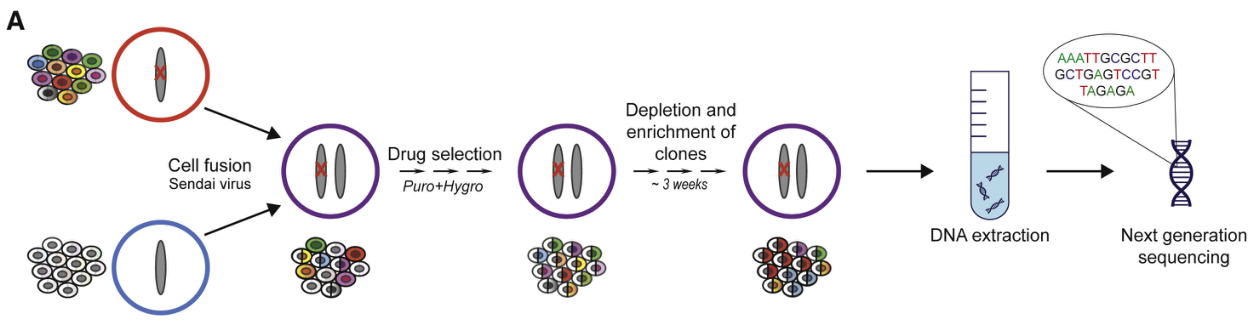
图2. 基于CRISPR的单倍剂量不足基因鉴定流程
通过筛查,共筛选出650个比较重要的单倍剂量不足基因,包含之前已有的基因与新发现的基因,并且用于软件预测模型建立的基因列表也存在于650个基因中,表明此方法具有较高的可靠性(图3)。
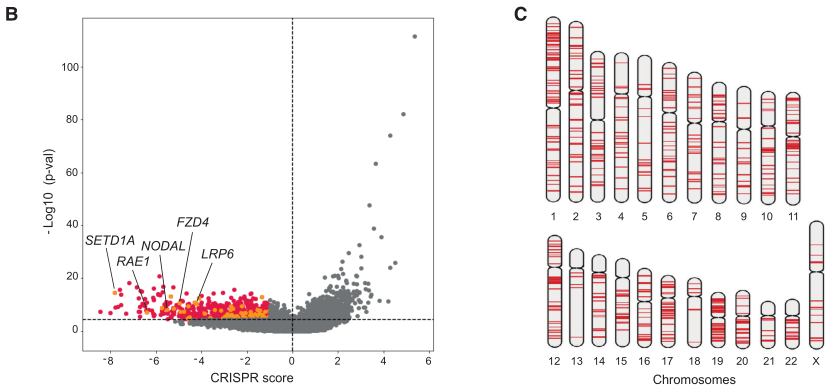
图3. 基于CRISPR方法筛选出来的650个单倍剂量不足基因
结论
单倍剂量不足现象是导致遗传病发生的一个原因,我们可以通过数据库查找、软件预测、高通量筛查的方法判断基因是否为单倍剂量不足,选择合适的方法可以对基因及其致病性进行解读,判断基因型对个体的影响。
参考文献
(1) Dang, V . T., Kassahn, K. S., Marcos, A. E. & Ragan, M. A. Identification of human haploinsufficient genes and their genomic proximity to segmental duplications. Eur J Hum Genet 16, 1350–1357 (2008).
(2) Huang, N., Lee, I., Marcotte, E. M. & Hurles, M. E. Characterising and predicting haploinsufficiency in the human genome. PLoS Genet 6, e1001154 (2010).
(3) Steinberg, J., Honti, F., Meader, S. & Webber, C. Haploinsufficiency predictions without study bias. Nucleic Acids Res 43, e101 (2015).
(4) Huang, N., Lee, I., Marcotte, E. M. & Hurles, M. E. Characterising and predicting haploinsufficiency in the human genome. PLoS Genet 6, e1001154 (2010).
(5) Steinberg, J., Honti, F., Meader, S. & Webber, C. Haploinsufficiency predictions without study bias. Nucleic Acids Res 43, e101 (2015).
(6) Han X , Chen S , Flynn E D , et al. Distinct Epigenomic Patterns Are Associated with Haploinsufficiency and Predict Risk Genes of Developmental Disorders[J]. Cold Spring Harbor Laboratory, 2017(1).
(7) Shihab HA, Rogers MF, Campbell C, Gaunt TR. HIPred: an integrative approach to predicting haploinsufficient genes. Bioinformatics. 2017 Jun 15;33(12):1751-1757.
(8) Sarel-Gallily R, Golan-Lev T, Yilmaz A, Sagi I, Benvenisty N. Genome-wide analysis of haploinsufficiency in human embryonic stem cells. Cell Rep. 2022 Mar 29;38(13):110573.